INNOVACIONES
Los métodos de secuenciación y ensamblaje de ARN pequeño revolucionan la detección de virus en la ciencia de cultivos y más
- Nutrición y seguridad alimentaria
- Reducción de la pobreza, medios de vida y empleo
Adopción o impacto a escala
Tomado por «próximos usuarios»
Inicio: 2007 | Fin: En curso
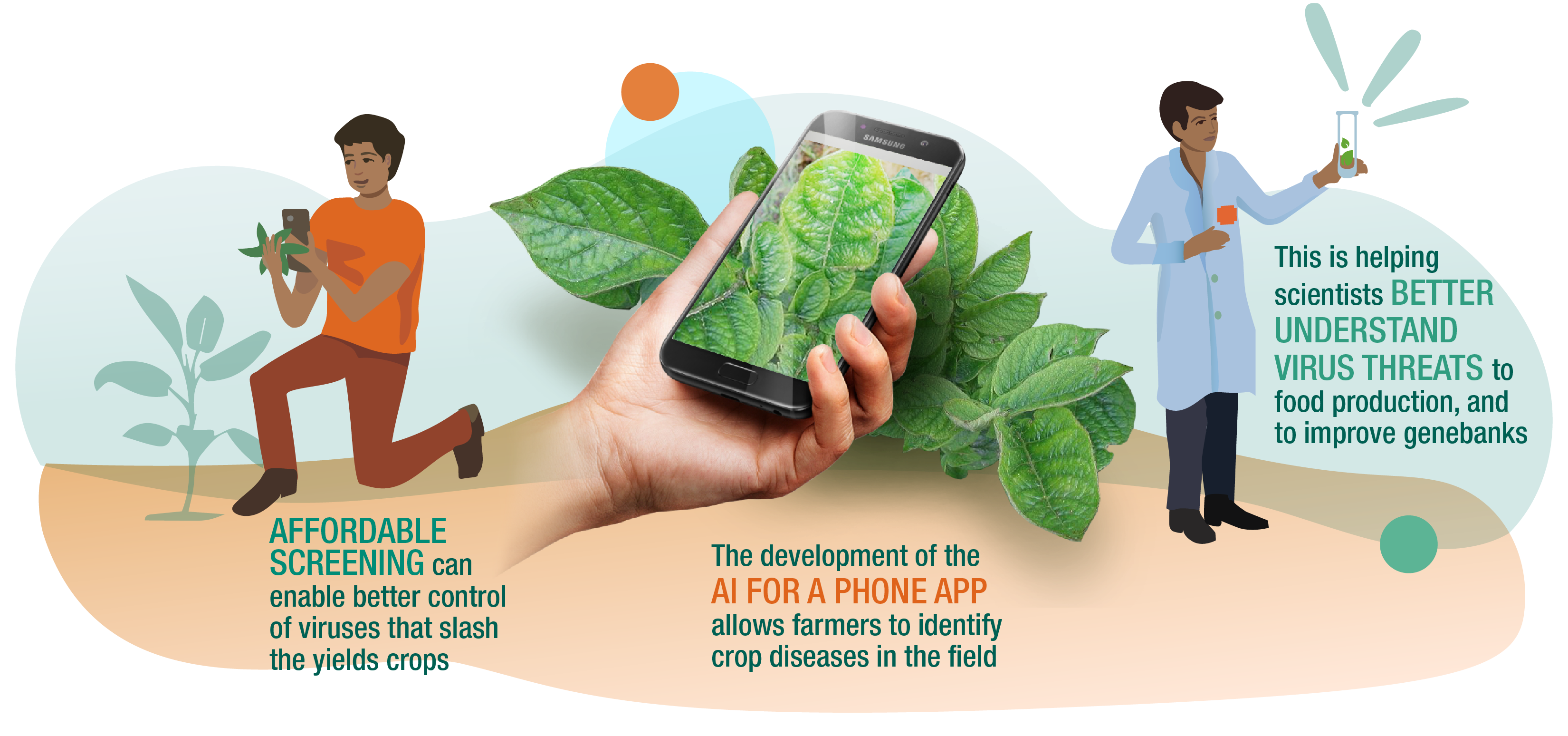
La detección eficiente y precisa de virus en plantas y animales es esencial para el desarrollo de estrategias efectivas para manejar la propagación y el impacto de las enfermedades. Los métodos de detección convencionales requieren conocimiento previo o información de secuencia de los patógenos potenciales y no son muy eficientes para detectar virus nuevos o variantes de virus. Un nuevo enfoque, el descubrimiento de virus mediante secuenciación de alta tasa de transferencia efectiva y ensamblaje de ARN pequeños totales (sRSA; Kreuze et al., 2009), ha demostrado ser altamente eficiente en la detección de virus de plantas y animales y ha sido ampliamente aprobado por los científicos y la comunidad de detección de enfermedades. Esto se ha mejorado mediante el uso de VirusDetect, una fuente de información de bioinformática automatizada que opera bajo Linux desarrollada por la Universidad de Cornell y CIP en 2017 que puede analizar de manera eficiente conjuntos de datos de ARN pequeño (ARNs) a gran escala para la identificación de virus nuevos y conocidos.

VirusDetect puede usarse para detectar virus en varios cultivos, hongos o animales. Ha sido adoptado por investigadores en varios países, incluidos algunos países africanos, y las unidades de salud de germoplasma del CGIAR lo están evaluando para su adopción como la herramienta estándar para certificar el estado libre de virus de cultivos clonales. Esos cultivos, que incluyen banano, yuca, papa y camote, sufren una gran pérdida de rendimiento debido a los virus porque los patógenos se propagan a través del material de siembra, así como a través de insectos vectores.
El uso de sRSA para detectar virus tiene un gran potencial para mejorar los esfuerzos y asegurar la salud de las plantas a nivel de bancos de germoplasma, en sistemas de semillas para cultivos de propagación vegetativa y en el campo.
Fundación Bill y Melinda Gates; Unión Europea (7PM); Fundación Howard G. Buffett; Fundación Nacional de Ciencias (EE. UU.).
Programa de investigación del CGIAR sobre Raíces, Tubérculos y Bananos; Instituto Internacional de Agricultura Tropical; Alliance Bioversity International y CIAT; Instituto Boyce Thomson (software); Universidad de Helsinki.